1. Introduction: The Role of AONs in Gene Therapy
This paper presents a comprehensive set of guidelines for designing Antisense Oligonucleotides (AONs) with a focus on splice modulation. AONs are molecular tools used to modify gene expression by targeting specific mRNA sequences, either to:
- Degrade the mRNA (gene silencing).
- Block translation.
- Modify splicing (splice modulation).
This study focuses on using AONs to induce exon skipping in the dystrophin gene, a therapeutic strategy for treating Duchenne Muscular Dystrophy (DMD).
2. Key Factors Influencing AON Efficiency
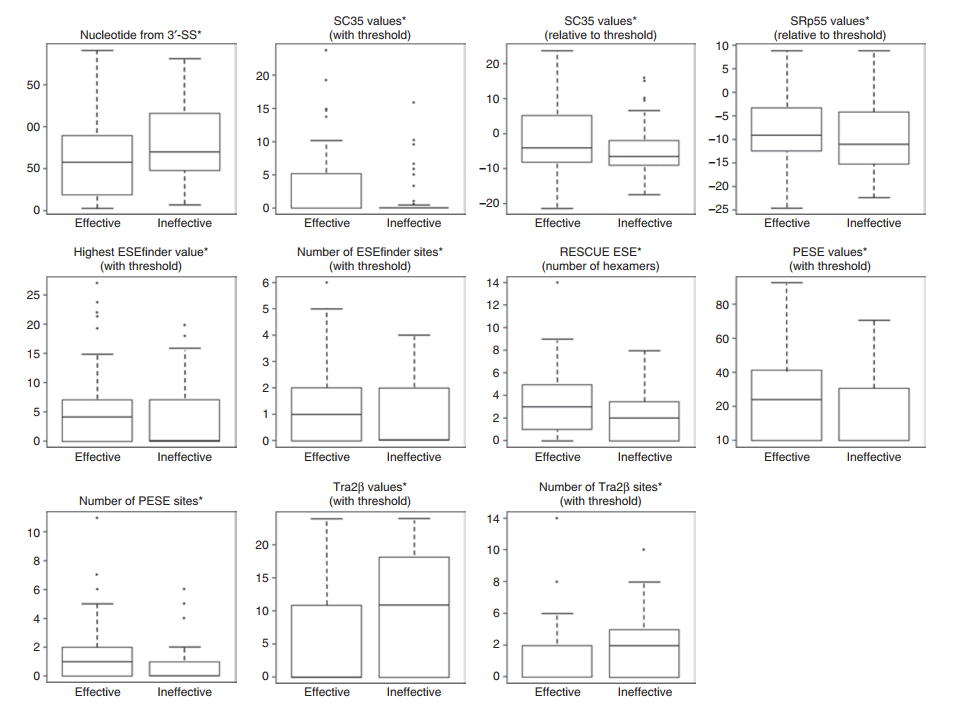
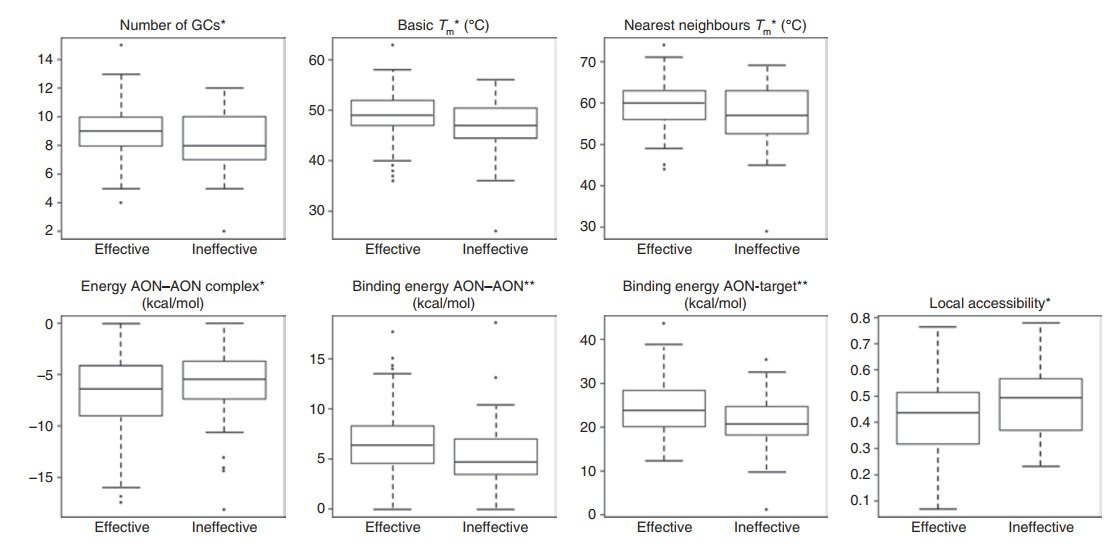
The study analyzed 156 AONs and classified them as effective or ineffective. The researchers identified several key factors that influence AON efficiency:
- Higher Guanine-Cytosine (GC) Content: AONs with higher GC content generally showed better efficiency.
- Binding Energy: AONs with stronger binding affinities to the target sequence were more effective.
- Location of AON Binding: Effective AONs typically bound near the acceptor splice site.
- Splicing Enhancers: AONs targeting regions rich in Exonic Splicing Enhancers (ESEs) showed improved efficiency.
- Tra2β Motifs: AONs targeting Tra2β motifs exhibited lower efficiency.
3. Computational and Experimental Analysis
The authors used advanced bioinformatics tools to predict secondary structures, binding energies, and splicing motifs. The results demonstrated that a combination of:
- Thermodynamic Properties (binding energy stability).
- Sequence Composition (GC content and motif analysis).
- Location Relative to Splicing Sites (targeting key regions).
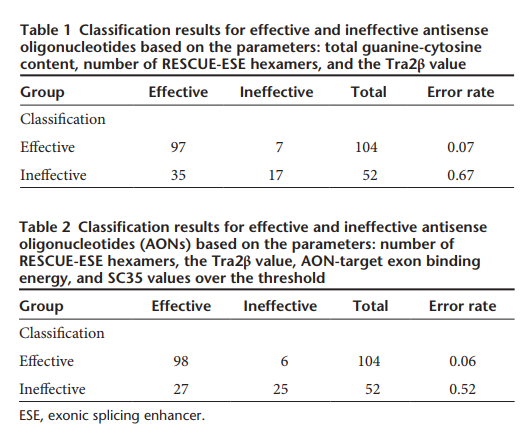
Using Linear Discriminant Analysis (LDA), the researchers successfully classified 79% of AONs based on these parameters, improving AON design accuracy.
4. Implications for Splice-Modulating Therapies
These findings provide valuable guidelines for designing splice-modulating AONs, which are critical for treating genetic diseases caused by splicing defects. The study also highlights:
- Predicting Secondary Structures: Essential for identifying stable binding regions.
- Considering Splicing Factors: SR proteins and Tra2β motifs play crucial roles in AON efficacy.
5. Conclusion: Toward More Effective AON Therapies
This research significantly improves AON design strategies by moving beyond trial-and-error approaches. By incorporating computational predictions and sequence-based optimizations, researchers can develop more efficient therapies for diseases like Duchenne Muscular Dystrophy (DMD).
The study underscores the need for continued refinement of AON selection based on thermodynamics, splicing motifs, and target site accessibility. These insights pave the way for more effective gene therapies for patients with splicing-related disorders.

| Published | 9/23/2008 |
| Address | doi:10.1038/mt.2008.205 |
| Authors | Annemieke Aartsma-Rus1, Laura van Vliet1, Marscha Hirschi1, Anneke AM Janson2, Hans Heemskerk1, Christa L de Winter1, Sjef de Kimpe2, Judith CT van Deutekom2, Peter AC ‘t Hoen1 and Gert-Jan B van Ommen1 |














