دیستروفی عضلانی دوشن و حذف اگزون ۵۳
دیستروفی عضلانی دوشن (DMD) یک اختلال ژنتیکی شدید است که به دلیل جهش در ژن دیستروفین ایجاد شده و منجر به تحلیل عضلانی و مرگ زودرس میگردد. الیگونوکلئوتیدهای آنتی سنس (AONs) درمانی امیدوارکننده هستند که با حذف اگزونهای معیوب ژن دیستروفین، تولید پروتئین عملکردی را بازمیگردانند.
ارزیابی حذف اگزون ۵۳ و بازسازی دیستروفین
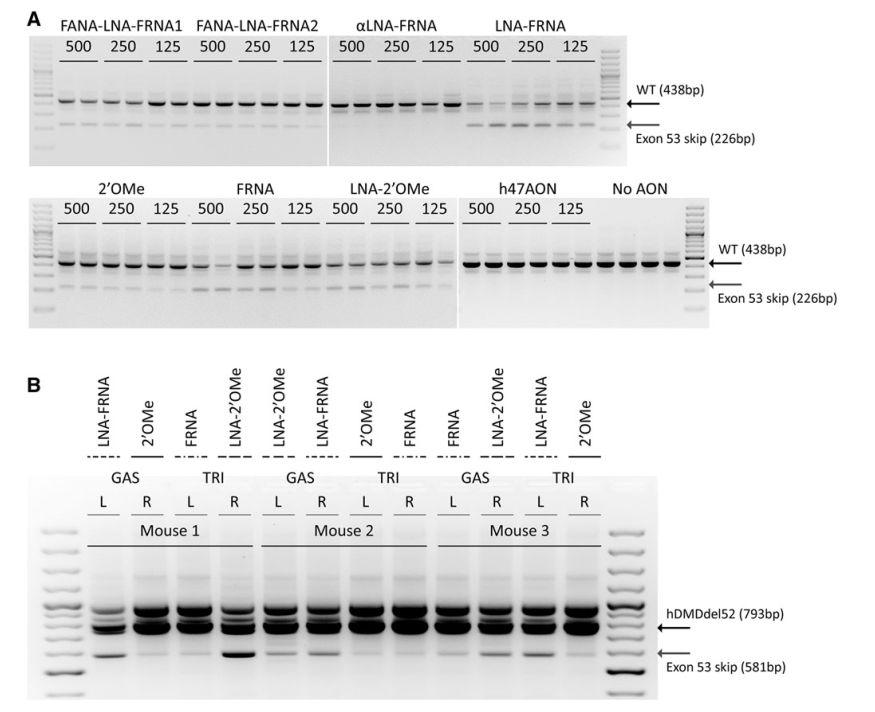
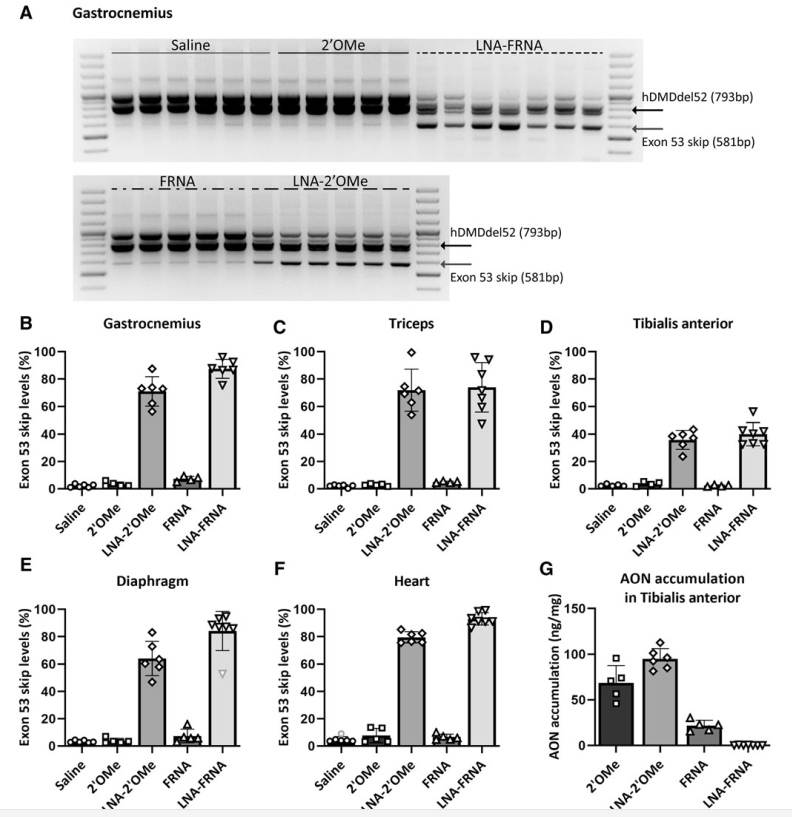
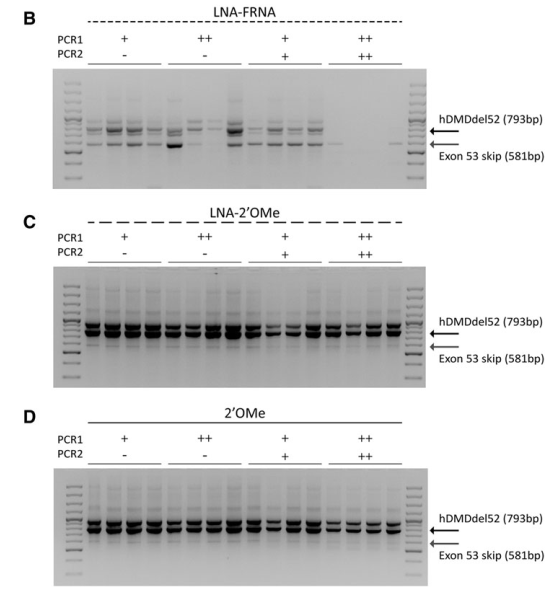
در این مطالعه، کارایی AONهایی که اگزون ۵۳ ژن دیستروفین را هدف قرار میدهند، در یک مدل موشی ارزیابی شده است. همچنین تغییرات شیمیایی برای افزایش حذف اگزون و بازسازی پروتئین مورد بررسی قرار گرفت. مقایسه AONها شامل 2′-O-methyl و LNA-FRNA در کشتهای میوبلاست انسانی و مدل موشی خاص (hDMDdel52/mdx) انجام شد. تزریقهای سیستمی و موضعی نیز برای بررسی کارایی این روش استفاده شد.
نتایج کلیدی مطالعه
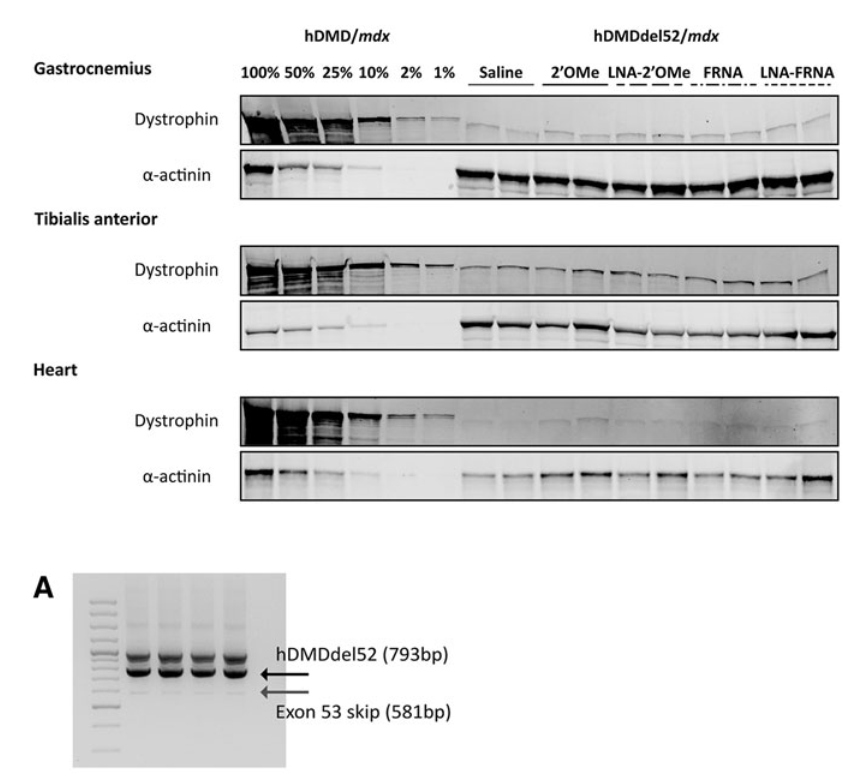
- حذف اگزون در برخی از AONها مانند LNA-FRNA و LNA-2′OMe بسیار بالا بود، اما بازسازی دیستروفین به میزان قابل توجهی پایینتر از انتظار باقی ماند.
- این اختلاف به دلیل تداخل AONها در حین تحلیل RNA بود که منجر به تخمین بالاتر از حد حذف اگزون شد.
- AONهای اصلاحشده با LNA اتصال قویتری به RNA نشان داده و باعث اختلال در سنتز cDNA شدند.
- برای کاهش این خطاها، پیشنهاد شد که دمای دناتوراسیون RNA به ۹۵ درجه سانتیگراد افزایش یابد.
نتیجهگیری و پیشنهادات آینده
AONهای اصلاحشده شیمیایی، به ویژه با تغییرات LNA، کارایی حذف اگزون را افزایش دادند، اما برای ارزیابی دقیقتر نتایج درمانی، نیاز به بررسی بیشتری وجود دارد. بهینهسازی طراحی AON و بررسی اهداف جایگزین اگزون برای افزایش اثربخشی درمانی در بیماران DMD توصیه میشود.









| Field | Details |
| Authors | Sarah Engelbeen, Daniel O’Reilly, Davy Van De Vijver, Ingrid Verhaart, Maaike van Putten, Vignesh Hariharan, Matthew Hassler, Anastasia Khvorova, Masad J. Damha, Annemieke Aartsma-Rus |
| Corresponding Author | Masad J. Damha, Annemieke Aartsma-Rus |
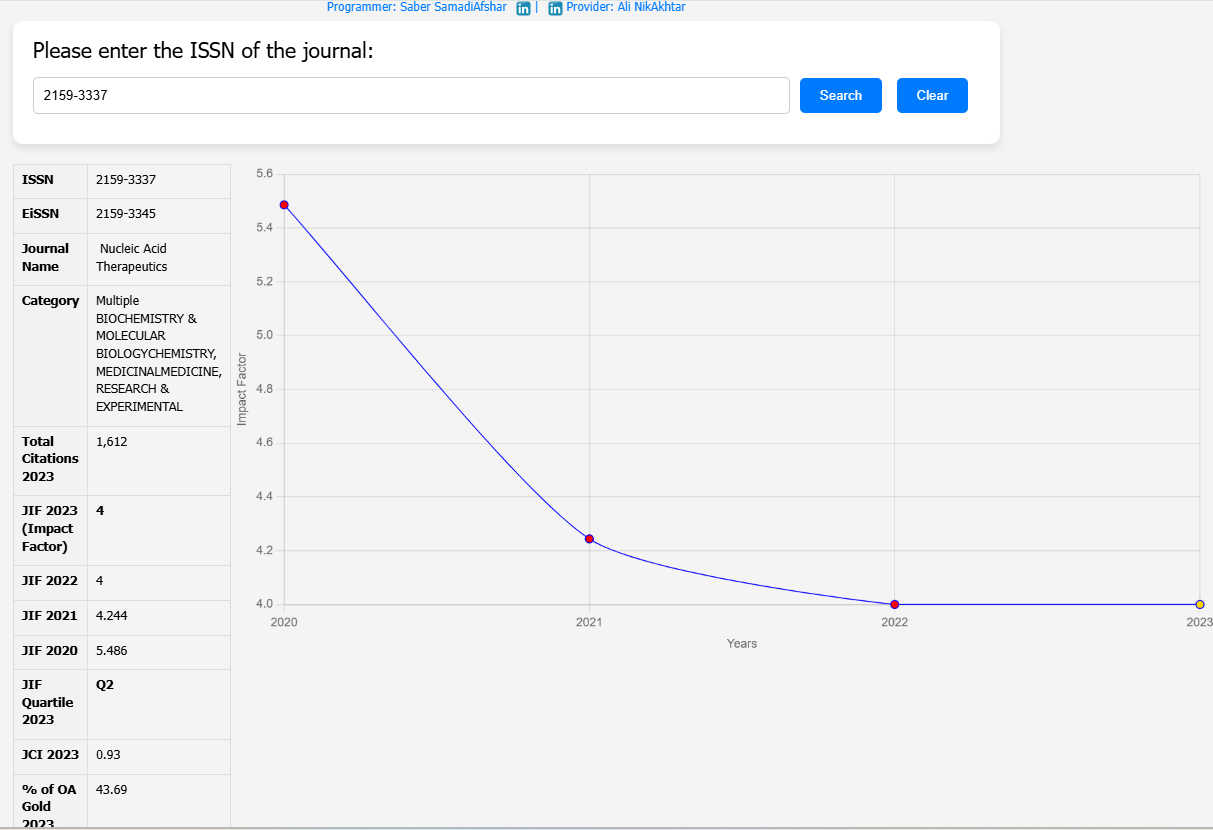
| Article Title | Challenges of Assessing Exon 53 Skipping of the Human DMD Transcript with Locked Nucleic Acid-Modified Antisense Oligonucleotides in a Mouse Model for Duchenne Muscular Dystrophy |
| Publication Date | 15-Jul-05 |
| Journal Name | Nucleic Acid Therapeutics |
| Keywords | Efficacy, locked nucleic acid, LNA, preclinical studies, exon skipping |
| Methods Used | In vitro analysis, in vivo validation, RT-PCR, western blot, AON synthesis, qPCR |
| DOI | 10.1089/nat.2023.0038 |