مدلسازی محاسباتی برای بهینهسازی حذف اگزون در درمان دیستروفی عضلانی دوشن (DMD)
دیستروفی عضلانی دوشن (DMD) یک بیماری ژنتیکی شدید است که به دلیل جهش در ژن DMD ایجاد شده و منجر به از دست رفتن پروتئین دیستروفین میشود. یکی از روشهای درمانی نویدبخش، حذف اگزون است که با استفاده از الیگونوکلئوتیدهای آنتیسنس (AONs) به بازگرداندن قاب خوانش درست پروتئین دیستروفین کمک میکند. با این حال، انتخاب توالیهای بهینه برای این فرایند همچنان یک چالش محسوب میشود.
روش غربالگری محاسباتی
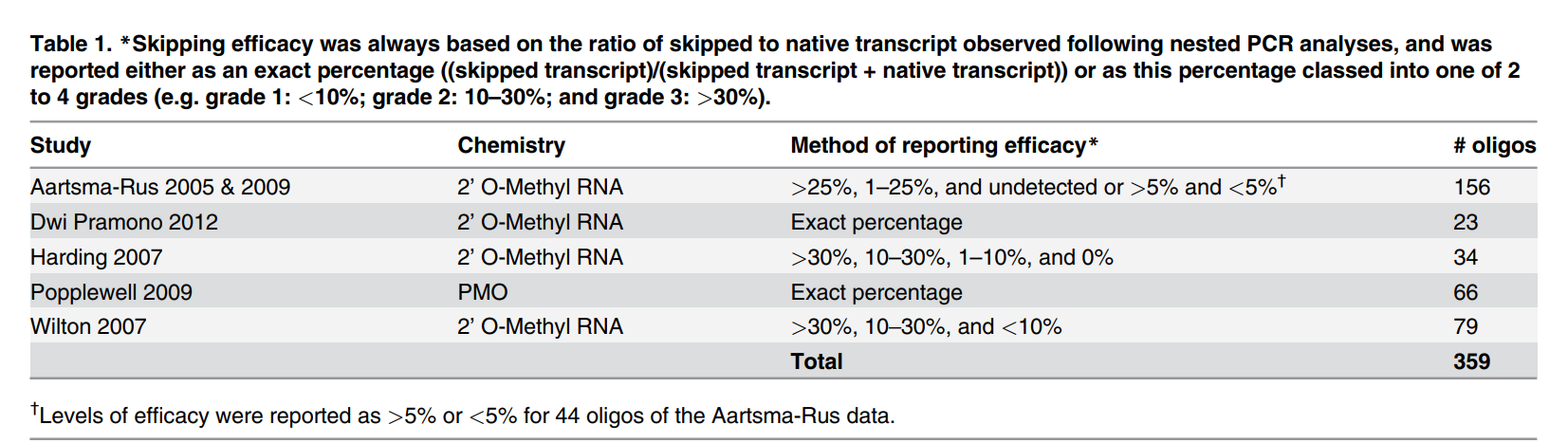
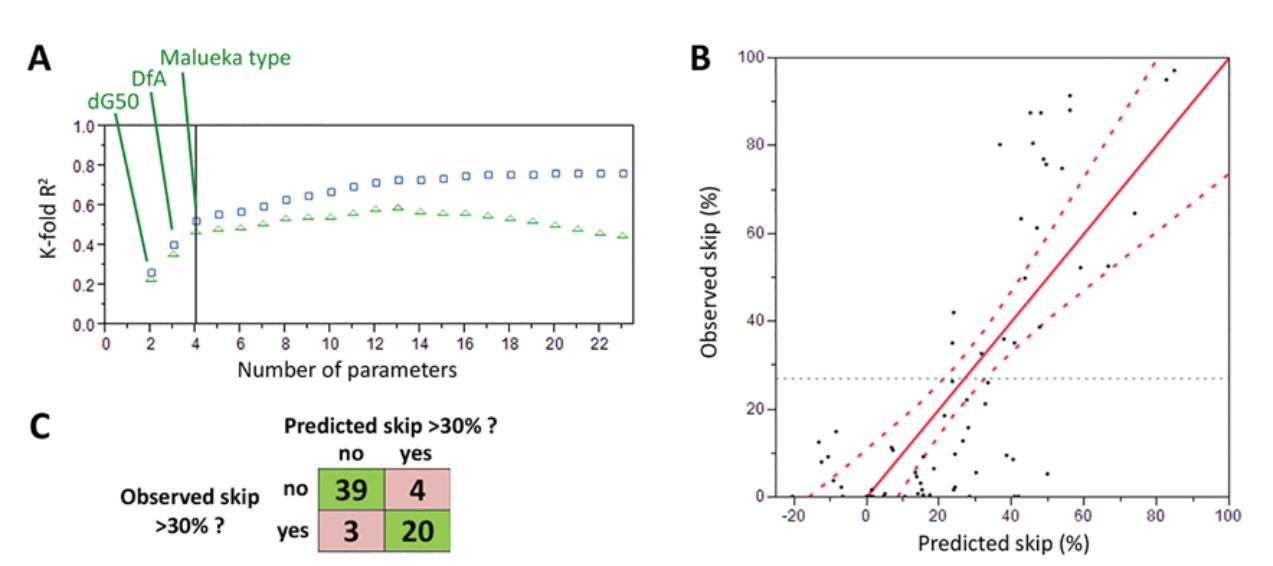
این مطالعه یک روش غربالگری محاسباتی (in silico) را معرفی میکند که از مدلسازی آماری پیشبینیکننده برای ارزیابی کارایی حذف اگزون استفاده میکند. با جمعآوری مجموعه دادههای گستردهای از الیگونوکلئوتیدهای آزمایششده قبلی، محققان دو پارامتر کلیدی را که بر حذف اگزون تأثیر میگذارند شناسایی کردند:
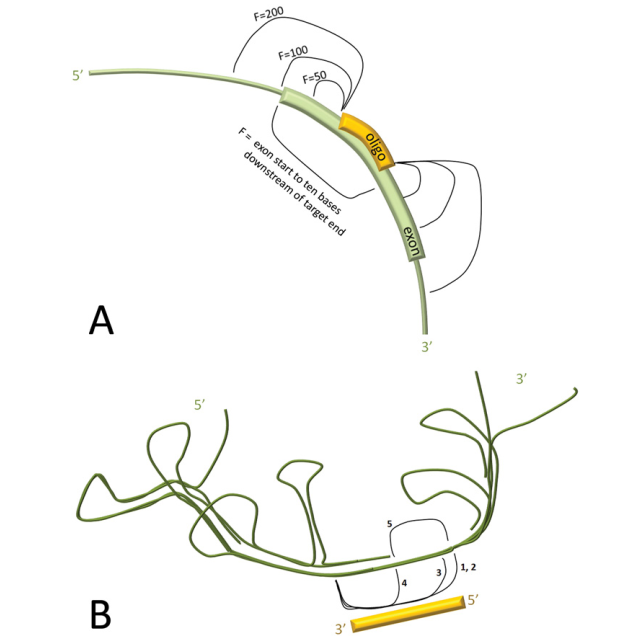
- انرژی اتصال الیگونوکلئوتید به RNA هدف
- فاصله محل هدف از جایگاه پذیرنده اگزون
نتایج مدلسازی و تایید آزمایشگاهی
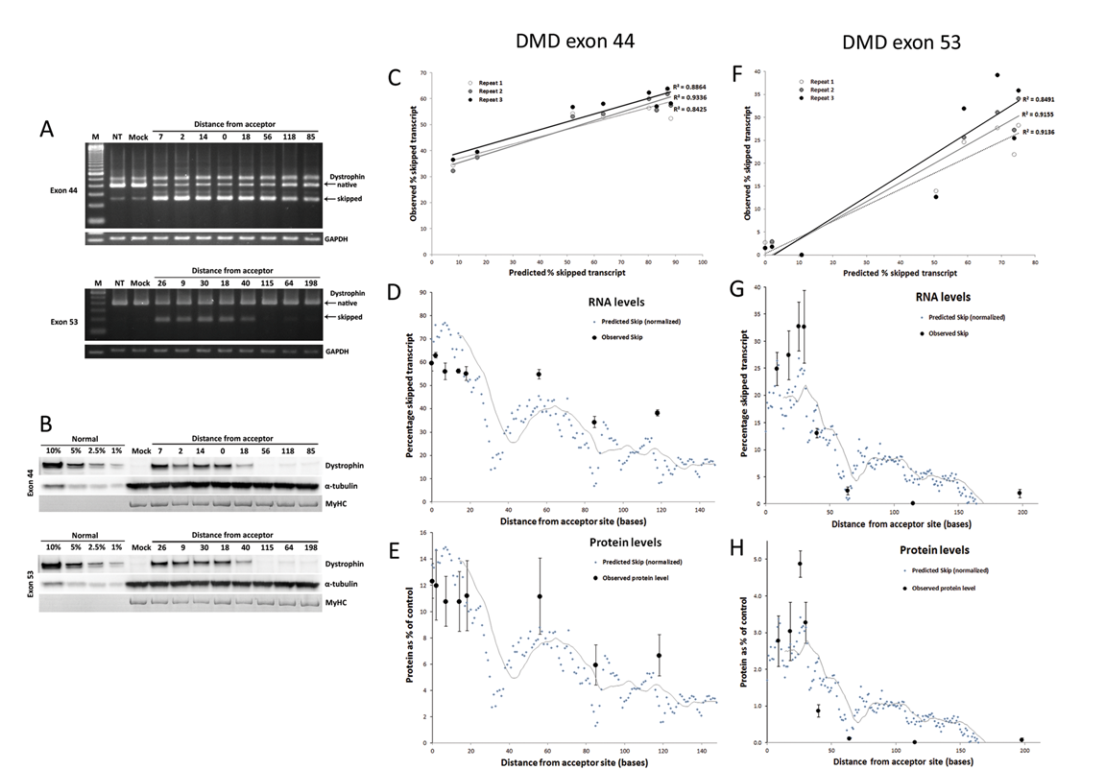
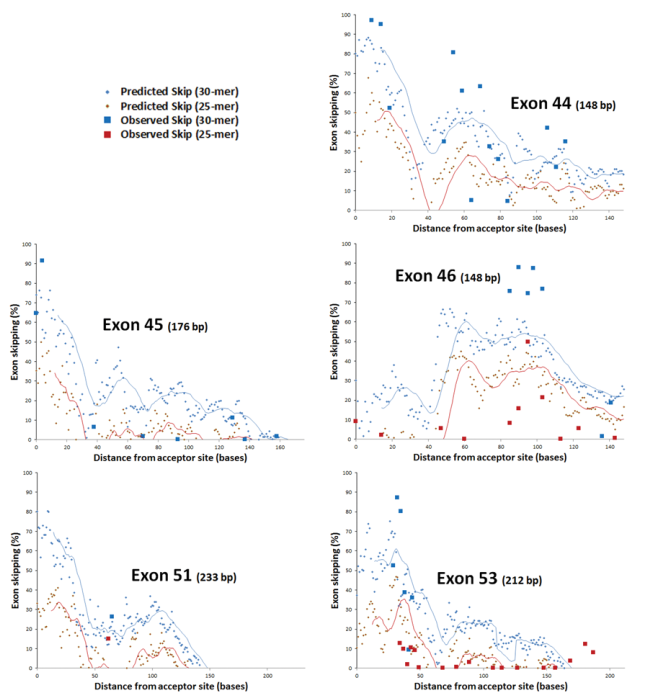
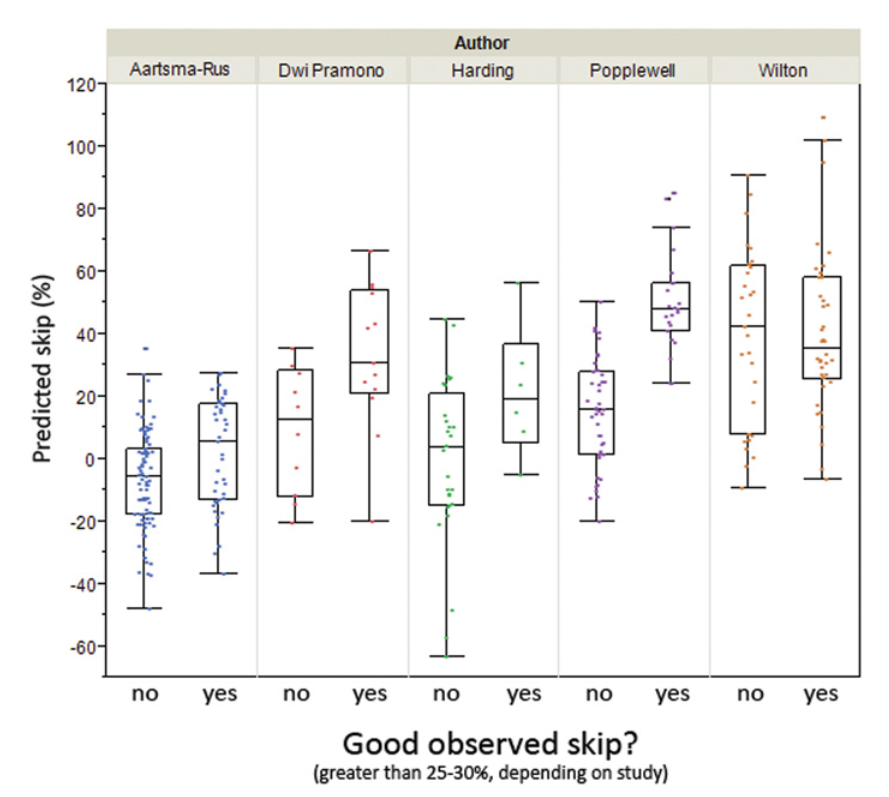
بر اساس این یافتهها، تیم تحقیقاتی الگوریتمهای پیشبینیکننده را توسعه داد که توانستند توالیهای با کارایی بالا و پایین را با دقت ۸۹٪ برای الیگونوکلئوتیدهای PMO و ۷۶٪ برای 2’-O-Methyl RNA طبقهبندی کنند. این پیشبینیها از طریق آزمایشهای حذف اگزون در شرایط آزمایشگاهی (in vitro) تأیید شدند که نشاندهنده همبستگی قوی با نتایج تجربی (R² = 0.89) بودند.
یافتههای کلیدی
- اثربخشی حذف اگزون با انرژی آزاد اتصال (ΔG) و موقعیت محل هدف ارتباط دارد.
- دستهبندی اگزون Malueka بر کارایی حذف اگزون در PMO تأثیر دارد اما در 2'-O-Methyl RNA تأثیری ندارد.
- یک ابزار غربالگری محاسباتی توسعه یافته است که پیشبینی حذف اگزون را در هر موقعیت هدفی امکانپذیر میکند.
نتیجهگیری
این مطالعه یک چارچوب محاسباتی برای طراحی منطقی الیگونوکلئوتیدها ارائه میدهد که باعث بهبود کارایی استراتژیهای حذف اگزون در DMD و سایر اختلالات ژنتیکی میشود.






| Field | Details |
| Title | In Silico Screening Based on Predictive Algorithms as a Design Tool for Exon Skipping Oligonucleotides in Duchenne Muscular Dystrophy |
| Authors | Yusuke Echigoya, Vincent Mouly, Luis Garcia, Toshifumi Yokota, William Duddy |
| Corresponding Author | William Duddy, Toshifumi Yokota |
| Publication Date | 27-Mar-15 |
| Journal | PLOS ONE |
| Keywords | Exon skipping, Duchenne muscular dystrophy, antisense oligonucleotides, in silico screening, predictive algorithms |
| Methods Used | Predictive statistical modeling, computational analysis, in vitro validation, RT-PCR, Western blot |
| Study Type | Research article |
| DOI | 10.1371/journal.pone.0120058 |